转自:华大基因
(1)靶点全覆盖|精准治疗靶点无遗漏
华择安mini非小细胞肺癌55基因检测不仅全面覆盖FDA/NMPA批准和NCCN/CSCO指南推荐的非小细胞肺癌靶向治疗相关靶点检测,还包含更多非小细胞肺癌潜在用药靶点基因、靶向治疗耐药相关基因和免疫治疗疗效预测生物标志物检测。
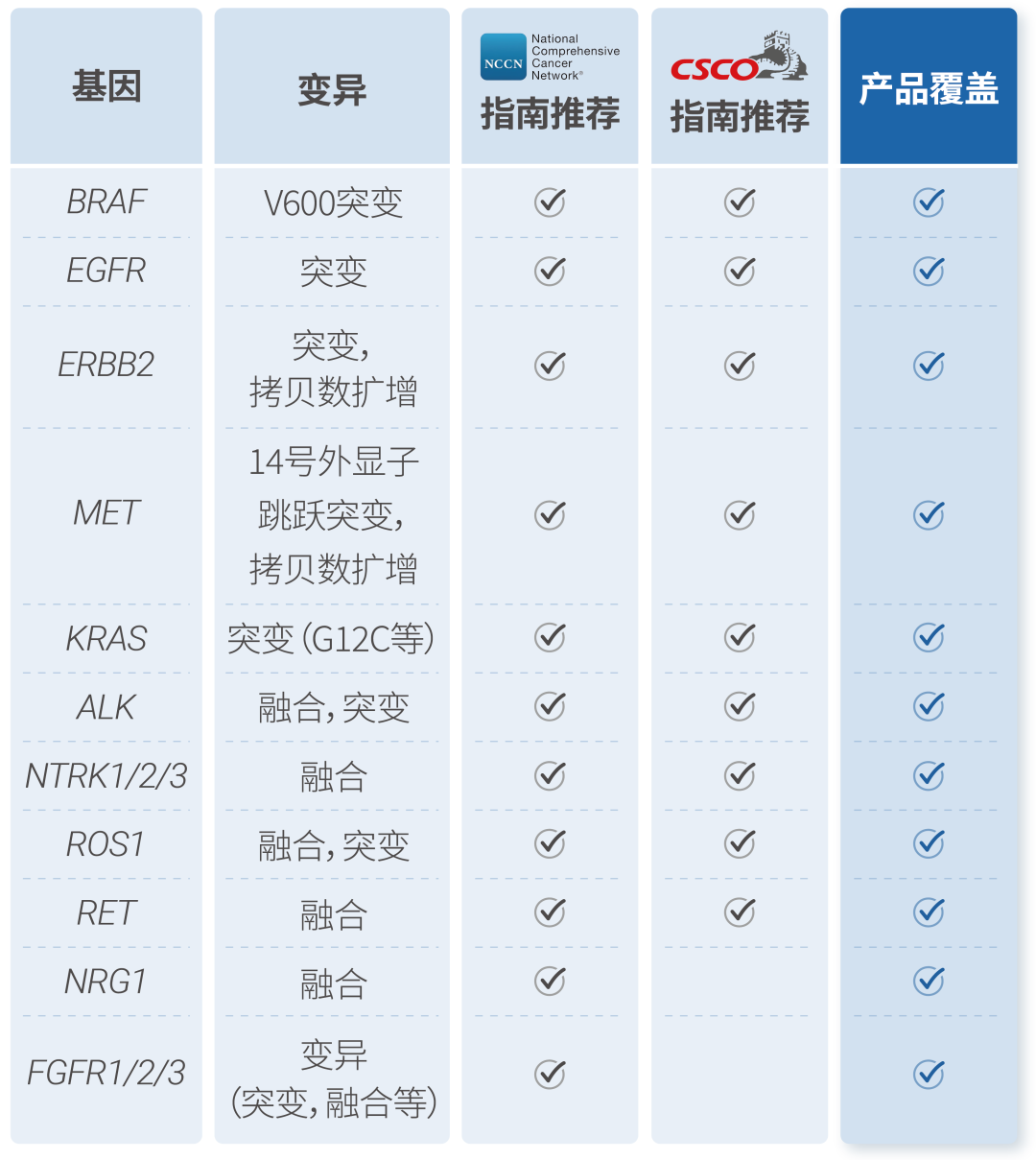
非小细胞肺癌国内外指南
推荐检测基因变异和产品覆盖情况
(2)检测精准锁定|DNA+RNA双组学共检,提升阳性检出率
DNA检测能够发现基因组层面的变异,包括SNV、Indel、CNV等类型;而RNA检测则能够直接反映基因的转录情况,尤其在识别融合变异上更具优势。DNA+RNA共检产品能够显著提高检测的灵敏度和准确性,提供最全面的基因变异信息,避免潜在的治疗靶点遗漏。
华择安mini非小细胞肺癌55基因检测通过DNA+RNA双组学共检,在识别融合变异上具备独特优势,有效提高融合变异检出。
10%~15%的NSCLC患者携带基因融合变异,提示可从靶向治疗中获益。
专家共识
建议晚期NSCLC患者应检测融合基因变异,为治疗策略的制定提供依据。其中ALK、ROS1、RET、NTRK作为融合变异检测的必检基因,NRG、FGFR、MET、EGFR、HER2(ERBB2)、BRAF作为扩展基因,本产品均全面覆盖。
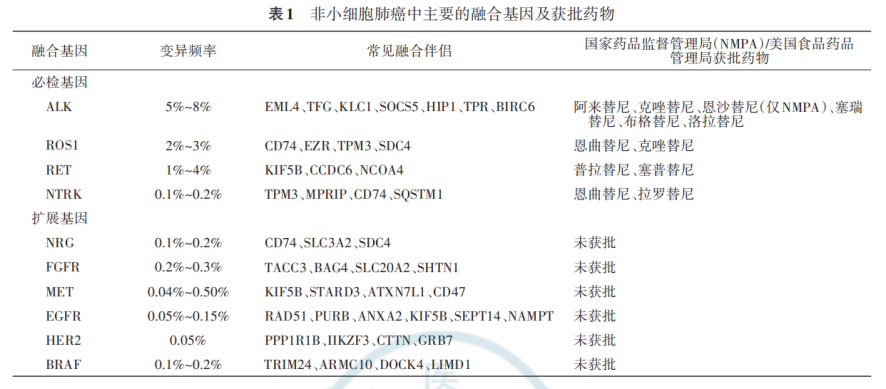
《非小细胞肺癌融合基因检测临床实践中国专家共识(2023版)》推荐检测融合基因及国内外获批药物
虽然DNA-basedNGS检测价值目前已经获得国内外指南和专家共识广泛认可,但在融合变异方面存在技术局限。
一方面是由于融合变异的断点通常位于冗长且富含重复序列的内含子区域,这些区域的GC含量变化不利于探针均匀有效地捕获,并且内部重复序列非常相似,也影响序列对比的准确性;另一方面,融合基因后续还要经历复杂的转录或转录后的剪接加工,这可能会改变融合蛋白功能,最终影响检出结果。
而与DNA-basedNGS相比,RNA-basedNGS方法可不受内含子区域干扰、直接在转录水平验证融合变异,对低丰度融合基因的检测能力更强,在识别新融合基因和变异形式方面具有优势,正好弥补了DNA-basedNGS不足,发现单DNA检测可能遗漏的融合靶点。双组学联合可以更大限度提升融合检出率,更精准指导临床用药。

国际权威指南和国内专家共识推荐RNA-basedNGS检测融合变异
临床研究
前期临床研究表明,在DNA-basedNGS检测为驱动基因变异阴性的232例肺腺癌患者中,经RNA-basedNGS可检测新发现14.2%的融合变异[3],并且其中80%患者接受对应靶向治疗可实现临床获益。
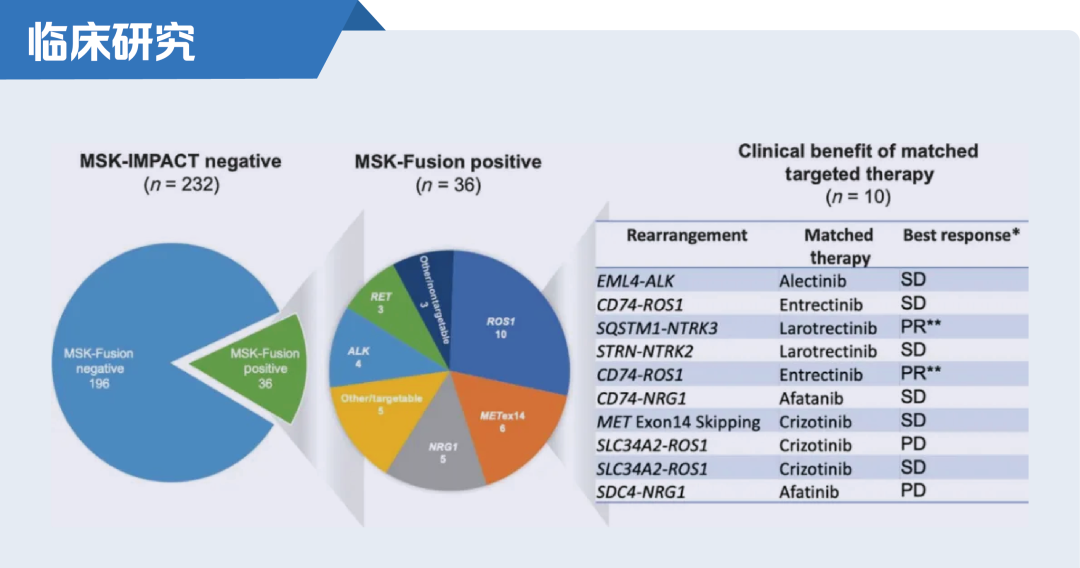
RNA层面检测的融合变异检出率
融合基因参与转录和翻译过程并最终表达出有功能的融合蛋白是患者治疗获益的关键。
一项回顾性研究表明,由DNA-basedNGS检出ALK或ROS1基因融合变异,而RNA-basedNGS和IHC均未检出的NSCLC患者最终未能从克唑替尼靶向治疗中获益[4]。
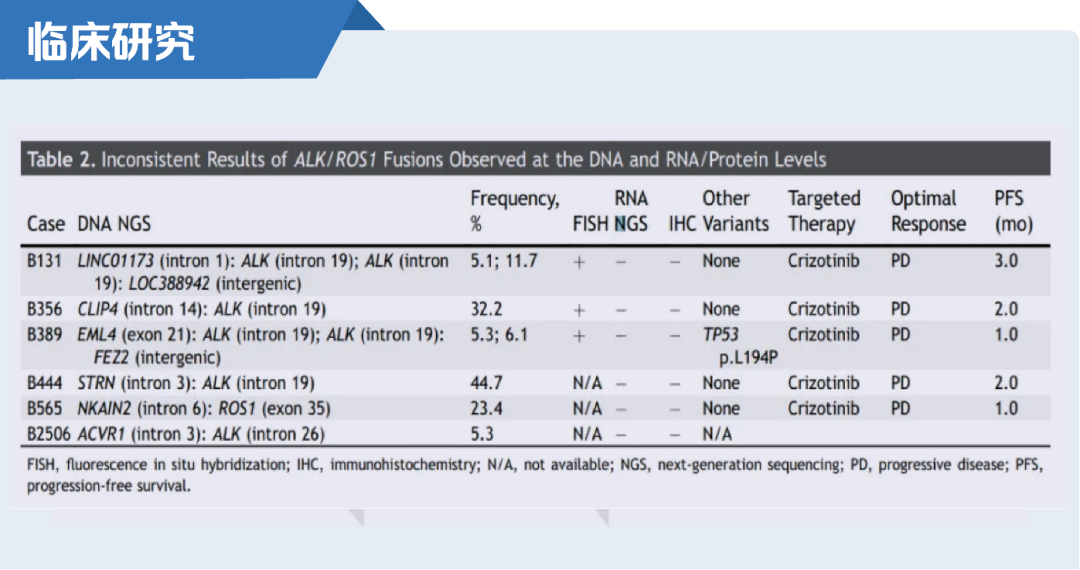
DNA-basedNGS阳性而RNA-basedNGS阴性患者,克唑替尼治疗未获益
(3)临床可及性强|节约送检样本,及时指导用药
运用“DNA+RNA共检”技术,一份组织样本实现DNA和RNA共检,更加节约临床样本,也为部分难以获取充足肿瘤组织样本的患者提供送检机会。一次完成DNA和RNA的检测,大大缩短了检测时间,为患者争取到了宝贵的治疗时机。
华大基因一直致力于为患者提供精准、全面的基因检测服务。
华大基因华择安实体瘤用药基因检测已上新!除华择安mini非小细胞肺癌用药55基因检测外,系列新品覆盖胃肠、肝胆胰、泌尿系统、乳腺、卵巢及子宫内膜癌等9大高发癌种,全面助力精准诊疗。
此外,华大基因还有华泛安®实体瘤全景用药基因检测及华见微®MRD定制化检测,结合先进的测序技术和专业的解读分析,为肺癌患者精准治疗提供有力支持!

参考资料:(滚动查看)
[1]Globalcancerstatistics2022:GLOBOCANestimatesofincidenceandmortalityworldwidefor36cancersin185countries-Bray-CA:ACancerJournalforClinicians-WileyOnlineLibrary.
https://acsjournals.onlinelibrary.wiley.com/doi/10.3322/caac.21834
[2]中华医学会肿瘤学分会.中华医学会肺癌临床诊疗指南患者版(2024版)[J].中华肿瘤杂志,2025,47(1):26-38.DOI:10.3760/cma.j.cn112152-20241024-00464
[3]BenayedR,OffinM,MullaneyK,etal.HighYieldofRNASequencingforTargetableKinaseFusionsinLungAdenocarcinomaswithNoMitogenicDriverAlterationDetectedbyDNASequencingandLowTumorMutationBurden.ClinCancerRes.2019;25(15):4712-4722.doi:10.1158/1078-0432.CCR-19-0225.
[4]LiW,GuoL,LiuY,etal.PotentialUnreliabilityofUncommonALK,ROS1,andRETGenomicBreakpointsinPredictingtheEfficacyofTargetedTherapyinNSCLC.JThoracOncol.2021;16(3):404-418.doi:10.1016/j.jtho.2020.10.156
(转自:华大基因)